11 Redes neuronales
A este tipo de algorimos se les suele llamar de “caja negra”. La caja negra se debe a que los modelos subyacentes se basan en sistemas matemáticos complejos y los resultados son difíciles de interpretar.
Aunque puede no ser factible interpretar los modelos de caja negra, es peligroso aplicar los métodos a ciegas.
11.1 Entendiendo una red neuronal
Una Red Neural Artificial (ANN) modela la relación entre un conjunto de señales de entrada y una señal de salida.
ANN usa una red de neuronas o nodos artificiales para resolver problemas de aprendizaje.
En términos generales, las RNA (ANN) son aprendices versátiles que se pueden aplicar a casi cualquier tarea de aprendizaje: clasificación, predicción numérica e incluso reconocimiento de patrones no supervisados.
Las RNA se aplican mejor a problemas donde los datos de entrada y los datos de salida son bien entendidos o al menos bastante simples, sin embargo, el proceso que relaciona la entrada con la salida es extremadamente complejo. Como método de caja negra, funcionan bien para este tipo de problemas de caja negra.
El diagrama de red dirigida define una relación entre las señales de entrada recibidas por los nodos (variables \(x\)) y la señal de salida (variable \(y\)).
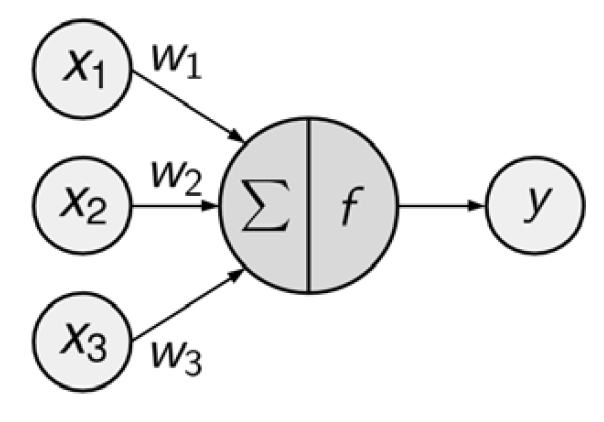
La señal de cada nodo se pondera (valores \(w\)) según su importancia; ignore por ahora cómo se determinan estos pesos. Las señales de entrada son sumadas por el cuerpo de la célula y la señal se transmite de acuerdo con una función de activación indicada por \(f\).
Una neurona artificial típica con \(n\) nodos de entrada puede representarse mediante la siguiente fórmula. Los pesos \(w\) permiten que cada una de las \(n\) entradas, (\(x\)), contribuya una cantidad mayor o menor a la suma de las señales de entrada. El total neto es utilizado por la función de activación \(f(x)\), y la señal resultante, \(y(x)\), es la salida.
\[ y(x) = f\left(\sum_i^nw_ix_i\right) \]
Aunque existen numerosas variantes de redes neuronales, cada una se puede definir en términos de las siguientes características:
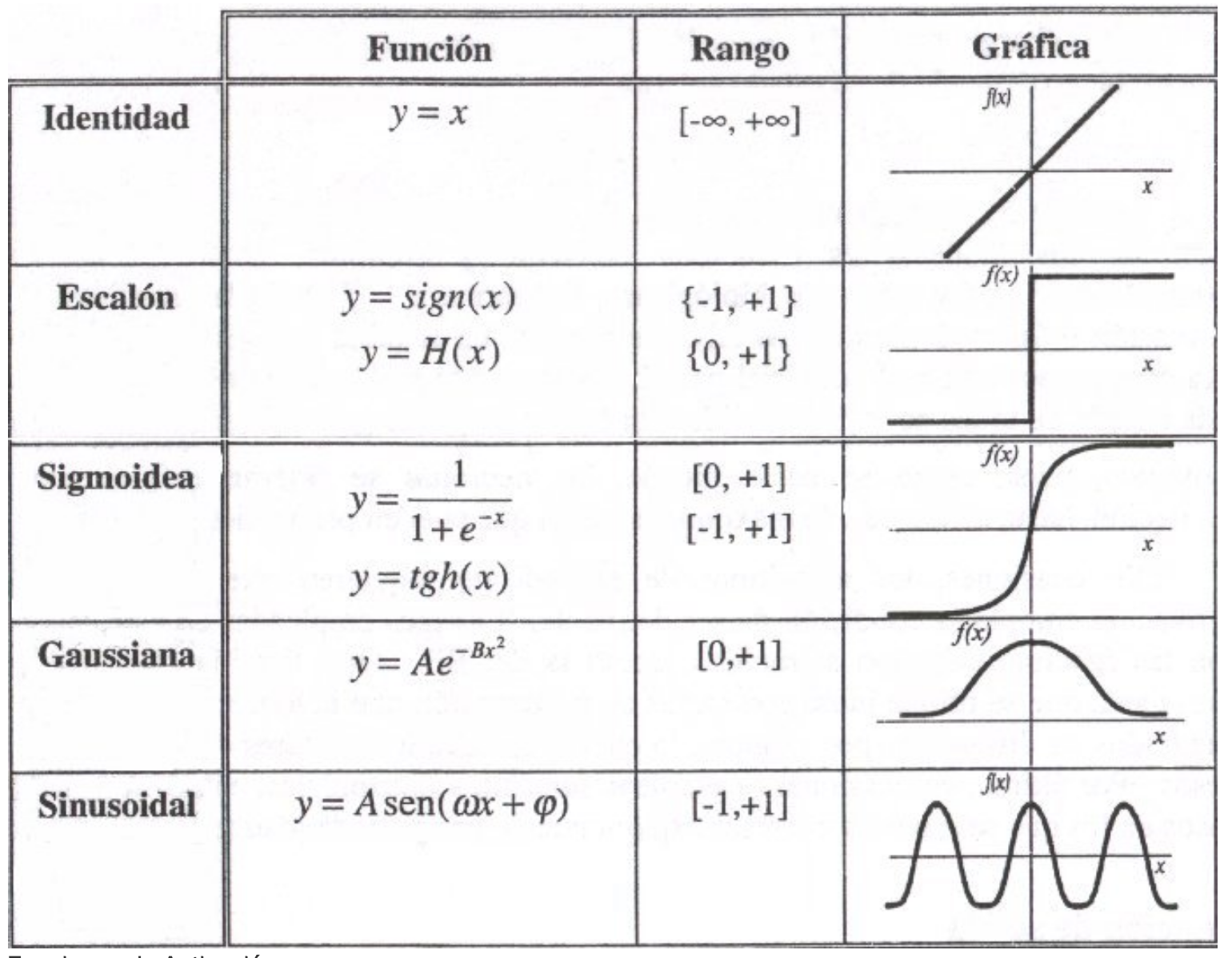
- Una función de activación, que transforma la señal de entrada neta de una neurona en una sola señal de salida para ser transmitida en la red
 {width=60% height = 80%}
{width=60% height = 80%}
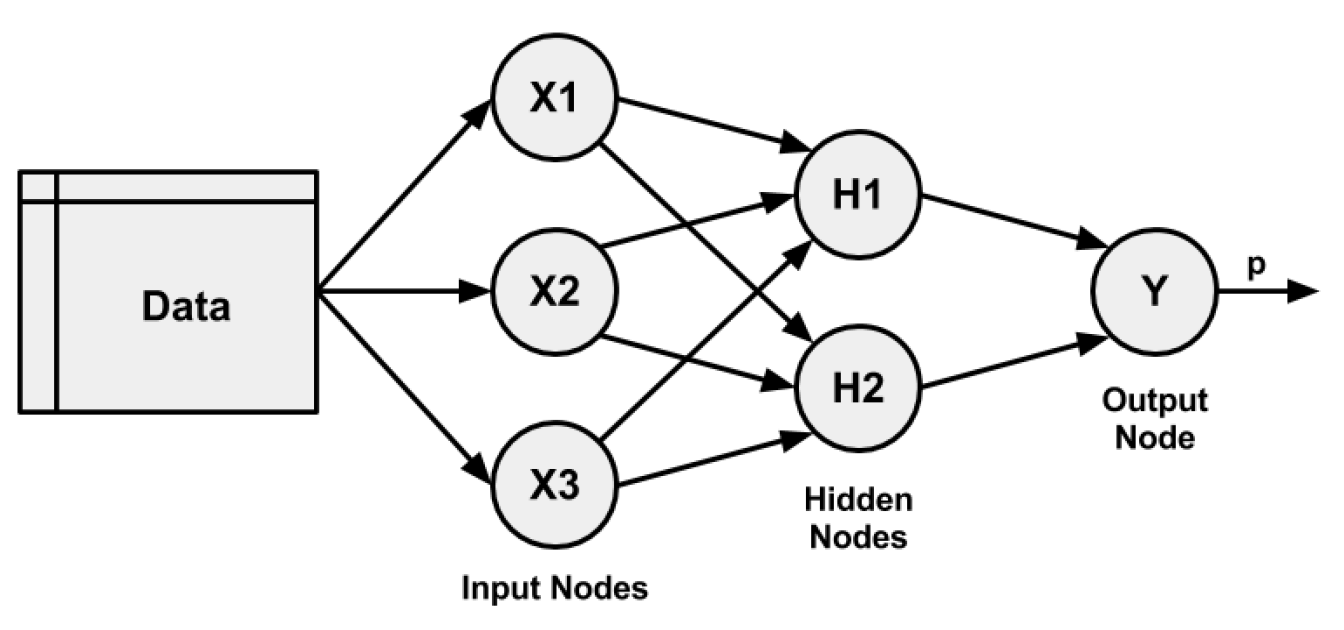
- Una topología de red (o arquitectura), que describe el número de neuronas en el modelo, así como el número de capas y la forma en que están conectadas.
 {width=60% height = 80%}
{width=60% height = 80%}
- El algoritmo de entrenamiento que especifica cómo se establecen los pesos de conexión para activar las neuronas en proporción a la señal de entrada.
| Fortalezas | Debilidades |
|---|---|
| Se puede adaptar a problemas de clasificación o predicción numérica | Reputación de ser computacionalmente intensivo y lento de entrenar, particularmente si la topología de red es compleja |
| Entre los enfoques de modelado más precisos | Datos de entrenamiento fáciles de sobreestimar o no subestmiar |
| Hace pocas suposiciones sobre las relaciones subyacentes de los datos | Resultados en un modelo complejo de caja negra que es difícil, si no imposible, de interpretar |
11.2 Paso 1: recopilación de datos
El conjunto de datos concretos contiene \(1030\) muestras de hormigón, con ocho características que describen los componentes utilizados en la mezcla. Se cree que estas características están relacionadas con la resistencia a la compresión final e incluyen la cantidad (en kilogramos por metro cúbico) de cemento, escoria, cenizas, agua, superplastificante, agregado grueso y agregado fino utilizado en el producto, además de el tiempo de envejecimiento (medido en días).
11.3 Paso 2: Explorar y preparar los datos
## 'data.frame': 1030 obs. of 9 variables:
## $ cement : num 141 169 250 266 155 ...
## $ slag : num 212 42.2 0 114 183.4 ...
## $ ash : num 0 124.3 95.7 0 0 ...
## $ water : num 204 158 187 228 193 ...
## $ superplastic: num 0 10.8 5.5 0 9.1 0 0 6.4 0 9 ...
## $ coarseagg : num 972 1081 957 932 1047 ...
## $ fineagg : num 748 796 861 670 697 ...
## $ age : int 28 14 28 28 28 90 7 56 28 28 ...
## $ strength : num 29.9 23.5 29.2 45.9 18.3 ...Las nueve variables en los datos corresponden a las ocho características y un resultado, aunque se ha evidenciado un problema. Las redes neuronales funcionan mejor cuando los datos de entrada se escalan a un rango estrecho alrededor de cero, y aquí vemos valores que van desde cero hasta más de mil.
Normalmente, la solución a este problema es reescalar los datos con una función de normalización o estandarización.
Si los datos siguen una curva en forma de campana (una distribución normal), entonces puede tener sentido utilizar la estandarización a través de la función
scale ()integrada de R.Por otro lado, si los datos siguen una distribución uniforme o son severamente no normales, entonces la normalización a un rango de 0-1 puede ser más apropiada.
normalize <- function(x) {
return((x - min(x)) / (max(x) - min(x)))
}
concrete_norm <- as.data.frame(lapply(concrete, normalize))
summary(concrete_norm$strength)## Min. 1st Qu. Median Mean 3rd Qu. Max.
## 0.0000 0.2664 0.4001 0.4172 0.5457 1.0000En comparación, los valores mínimos y máximos originales fueron 2.33 y 82.6
## Min. 1st Qu. Median Mean 3rd Qu. Max.
## 2.33 23.71 34.45 35.82 46.13 82.60Cualquier transformación aplicada a los datos antes de entrenar el modelo tendrá que aplicarse en reversa más adelante para volver a las unidades de medida originales. Para facilitar el cambio de escala, es conveniente guardar los datos originales, o al menos las estadísticas de resumen de los datos originales.
Dividiremos los datos en un conjunto de capacitación con el 75 por ciento de los ejemplos y un conjunto de prueba con el 25 por ciento.
11.4 Paso 3: entrenar un modelo en los datos
Para modelar la relación entre los ingredientes usados en el concreto y la resistencia del producto terminado, usaremos una red neuronal feedforward (red que no tiene ciclos) de múltiples capas. El paquete neuralnet (Fritsch and Guenther (2016)) proporciona una implementación estándar y fácil de usar de tales redes. También ofrece una función para trazar la topología de red (más paquetes aquí).
Empezaremos a entrenar la red con solo un nodo oculto
library(neuralnet)
concrete_model <- neuralnet(strength ~ cement + slag +
ash + water + superplastic +
coarseagg + fineagg + age,
data = concrete_train)Grafiquemos el resultado
En este modelo simple, hay un nodo de entrada para cada una de las ocho características, seguido de un único nodo oculto y un único nodo de salida que predice la fuerza del concreto.
Los pesos para cada una de las conexiones también se representan, al igual que los términos de sesgo (indicados por los nodos con un 1).
El diagrama también informa el número de pasos de entrenamiento y una medida llamada, la suma de los errores cuadrados (SSE). Estas métricas serán útiles cuando estemos evaluando el rendimiento del modelo.
En ocasiones es importante estimar la complejidad del modelo cuand trabajamos con más datos:
nn_test = function(data)
{
concrete_model <- neuralnet(strength ~ cement + slag +
ash + water + superplastic +
coarseagg + fineagg + age,
data = data)
}
# devtools::install_github("agenis/GuessCompx")
#library(GuessCompx) # get it by running: install.packages("GuessCompx")
library(neuralnet)
#CompEst(concrete_train, nn_test,plot =FALSE)11.5 Paso 4: evaluar el rendimiento del modelo
El diagrama de topología de red nos permite observar la caja negra de la ANN, pero no proporciona mucha información sobre qué tan bien el modelo se ajusta a nuestros datos. Para estimar el rendimiento de nuestro modelo, podemos usar la función predict() para generar predicciones en el conjunto de datos de prueba:
Ten en cuenta que la función de predict() funciona de manera un poco diferente de las funciones de predict() que hemos utilizado hasta ahora. Devuelve una lista con dos componentes: $neuronas, que almacena las neuronas para cada capa en la red, y $net.results, que almacena los valores predichos. Vamos a querer lo último:
Debido a que este es un problema de predicción numérica en lugar de un problema de clasificación, no podemos usar una matriz de confusión para examinar la precisión del modelo. En cambio, debemos medir la correlación entre nuestra fuerza del concreto predicha y el valor verdadero. Esto proporciona una idea de la fuerza de la asociación lineal entre las dos variables.
## [,1]
## [1,] 0.8059025No te alarmes si el resultado difiere. Debido a que la red neuronal comienza con pesos aleatorios, las predicciones pueden variar de un modelo a otro.
Si la correlación es alta implica que el modelo está haciendo un buen trabajo.
11.6 Paso 5: mejorando el ajuste
Como las redes con topologías más complejas son capaces de aprender conceptos más difíciles, veamos qué sucede cuando aumentamos la cantidad de nodos ocultos a cinco.
concrete_model2 <- neuralnet(strength ~ cement + slag +
ash + water + superplastic +
coarseagg + fineagg + age,
data = concrete_train, hidden = 5)Observa que el error informado (medido nuevamente por SSE) se ha reducido.
Comparemos los resultados:
predicted_strength2 <- predict(concrete_model2, concrete_test[1:8])
# predicted_strength2 <- model_results2$net.result
cor(predicted_strength2, concrete_test$strength)## [,1]
## [1,] 0.9364437denormalize <- function(x_norm,x_orig)
{
#x_norm: normalized dats
#x_orig: original data
mmin <- min(x_orig);mmax <- max(x_orig)
denorm_val <- x_norm*(mmax-mmin)+mmin
return(denorm_val)
}
library(caret)
R2(denormalize(predicted_strength2,concrete$strength),
denormalize(concrete_test$strength,concrete$strength))## [,1]
## [1,] 0.8769268Parámetros más importantes
hidden: vector de enteros con el número de neuronas escondidas. Ejemplo,hidden=c(4,3,2)indica que se tiene tres capas ocultas con 4,3, y 2 nodos respectivamente. Por defecto es igual a 1.act.fct: función de activiación. Se puede usarlogisticytanh, la primera es por defecto.err.fct: función de pérdida con la que se evalúa el error cometido en las estimación durante la estimación de los pesos \(w\). Por defecto es igual asse(errores al cuadrado).
Ajustemos un modelo con una arquitectura más compleja:
concrete_model3 <- neuralnet(strength ~ cement + slag +
ash + water + superplastic +
coarseagg + fineagg + age,
data = concrete_train, hidden = c(5,2,3),act.fct = "tanh")
plot(concrete_model3)
predicted_strength3 <- predict(concrete_model3, concrete_test[1:8])
R2(denormalize(predicted_strength3,concrete$strength),
denormalize(concrete_test$strength,concrete$strength))## [,1]
## [1,] 0.849131411.6.1 Usando caret
Como podemos ver, podemos variar los parámetros elegidos para elegir la mejor combinación de, por ejemplo, el número de capas y nodos. Podemos hacerlo con caret
tune.grid.neuralnet <- expand.grid(
layer1 = 2:3,
layer2 = 2:3,
layer3 = 2:3
)
control <- trainControl(method="repeatedcv", number=5)
nnet_caret <- caret::train(strength ~ cement + slag + ash + water + superplastic + coarseagg + fineagg + age,
data = concrete_train,
method = 'neuralnet',
linear.output = TRUE,
tuneGrid = tune.grid.neuralnet,
metric = "RMSE",
trControl = control)
nnet_caret$finalModel$tuneValue # valores de las capas## layer1 layer2 layer3
## 7 3 3 2predicted_strength4 <- predict(nnet_caret,concrete_test[1:8])
c(m1 =R2(denormalize(predicted_strength,concrete$strength),
denormalize(concrete_test$strength,concrete$strength))
,
m2 = R2(denormalize(predicted_strength2,concrete$strength),
denormalize(concrete_test$strength,concrete$strength))
,
m3 = R2(denormalize(predicted_strength3,concrete$strength),
denormalize(concrete_test$strength,concrete$strength))
,
m4 = R2(denormalize(predicted_strength4,concrete$strength),
denormalize(concrete_test$strength,concrete$strength)))## m1 m2 m3 m4
## 0.6494788 0.8769268 0.8491314 0.845043511.6.2 Ejercicio
Usa los datos Hitters (salarios de jugadores de baseball con estadísticas de su rendimiento) cargándolos con:
library(ISLR2)
library(caret)
set.seed(8519)
# 1) Datos y partición
Gitters <- na.omit(Hitters)
depvar <- "Salary"
X_in <- Gitters[, setdiff(names(Gitters), depvar)]
y_raw <- Gitters[[depvar]]
index <- createDataPartition(y_raw, p = 0.75, list = FALSE)
train_df <- X_in[index, , drop = FALSE]
test_df <- X_in[-index, , drop = FALSE]
y_train <- as.numeric(scale(y_raw[index])) # vector, no matriz
y_test <- as.numeric(scale(y_raw[-index]))
# 2) Dummies y preprocesado (ajustado SOLO con train)
dummies <- dummyVars(~ ., data = train_df, fullRank = TRUE)
X_train_dum <- predict(dummies, newdata = train_df)
X_test_dum <- predict(dummies, newdata = test_df)
# Quita columnas de varianza casi nula
nzv <- nearZeroVar(X_train_dum)
if (length(nzv)) {
X_train_dum <- X_train_dum[, -nzv, drop = FALSE]
X_test_dum <- X_test_dum[, -nzv, drop = FALSE]
}
# Center/scale SOLO con train
pp <- preProcess(X_train_dum, method = c("center", "scale"))
X_train <- as.data.frame(predict(pp, X_train_dum))
X_test <- as.data.frame(predict(pp, X_test_dum))
# 3) Entrenamiento con neuralnet (aumenta stepmax y usa rprop+)
tune.grid.neuralnet <- expand.grid(
layer1 = 2:3,
layer2 = 0:2, # permite capas vacías (más estable)
layer3 = 0 # solo 1–2 capas
)
ctrl <- trainControl(method = "repeatedcv", number = 5, repeats = 1)
set.seed(8519)
nnet_caret <- caret::train(
x = X_train, y = y_train,
method = "neuralnet",
tuneGrid = tune.grid.neuralnet,
trControl = ctrl,
metric = "RMSE",
linear.output = TRUE,
algorithm = "rprop+",
stepmax = 2e5,
lifesign = "minimal"
)## hidden: 2 thresh: 0.01 rep: 1/1 steps: 91133 error: 7.93512 time: 3.34 secs
## hidden: 3 thresh: 0.01 rep: 1/1 steps: 45601 error: 5.90109 time: 1.83 secs
## hidden: 2, 1 thresh: 0.01 rep: 1/1 steps: 4248 error: 18.47307 time: 0.2 secs
## hidden: 3, 1 thresh: 0.01 rep: 1/1 steps: 13401 error: 5.25173 time: 0.65 secs
## hidden: 2, 2 thresh: 0.01 rep: 1/1 steps: stepmax min thresh: 0.0123381654020742## Warning: Algorithm did not converge in 1 of 1 repetition(s) within the stepmax.## Warning: predictions failed for Fold1.Rep1: layer1=2, layer2=2, layer3=0 Error in cbind(1, pred) %*% weights[[num_hidden_layers + 1]] :
## requires numeric/complex matrix/vector arguments## hidden: 3, 2 thresh: 0.01 rep: 1/1 steps: 24070 error: 10.11108 time: 1.18 secs
## hidden: 2 thresh: 0.01 rep: 1/1 steps: 18170 error: 10.61841 time: 0.62 secs
## hidden: 3 thresh: 0.01 rep: 1/1 steps: 1451 error: 24.07298 time: 0.05 secs
## hidden: 2, 1 thresh: 0.01 rep: 1/1 steps: 5039 error: 15.05091 time: 0.22 secs
## hidden: 3, 1 thresh: 0.01 rep: 1/1 steps: 47523 error: 8.38524 time: 2.21 secs
## hidden: 2, 2 thresh: 0.01 rep: 1/1 steps: 30116 error: 17.59561 time: 1.27 secs
## hidden: 3, 2 thresh: 0.01 rep: 1/1 steps: 29069 error: 3.85045 time: 1.46 secs
## hidden: 2 thresh: 0.01 rep: 1/1 steps: 2029 error: 21.74723 time: 0.06 secs
## hidden: 3 thresh: 0.01 rep: 1/1 steps: 78015 error: 5.75582 time: 3.3 secs
## hidden: 2, 1 thresh: 0.01 rep: 1/1 steps: 27378 error: 12.32146 time: 1.21 secs
## hidden: 3, 1 thresh: 0.01 rep: 1/1 steps: 7125 error: 3.35723 time: 0.33 secs
## hidden: 2, 2 thresh: 0.01 rep: 1/1 steps: 68265 error: 6.76327 time: 3.01 secs
## hidden: 3, 2 thresh: 0.01 rep: 1/1 steps: 15877 error: 4.10099 time: 0.76 secs
## hidden: 2 thresh: 0.01 rep: 1/1 steps: 43210 error: 7.19069 time: 1.59 secs
## hidden: 3 thresh: 0.01 rep: 1/1 steps: 26663 error: 8.11192 time: 1.06 secs
## hidden: 2, 1 thresh: 0.01 rep: 1/1 steps: 12921 error: 13.43768 time: 0.55 secs
## hidden: 3, 1 thresh: 0.01 rep: 1/1 steps: 9839 error: 4.09715 time: 0.51 secs
## hidden: 2, 2 thresh: 0.01 rep: 1/1 steps: stepmax min thresh: 0.0135449698147733## Warning: Algorithm did not converge in 1 of 1 repetition(s) within the stepmax.## Warning: predictions failed for Fold4.Rep1: layer1=2, layer2=2, layer3=0 Error in cbind(1, pred) %*% weights[[num_hidden_layers + 1]] :
## requires numeric/complex matrix/vector arguments## hidden: 3, 2 thresh: 0.01 rep: 1/1 steps: 21009 error: 5.64973 time: 1.03 secs
## hidden: 2 thresh: 0.01 rep: 1/1 steps: 8030 error: 16.75476 time: 0.28 secs
## hidden: 3 thresh: 0.01 rep: 1/1 steps: 12082 error: 9.17427 time: 0.51 secs
## hidden: 2, 1 thresh: 0.01 rep: 1/1 steps: 10755 error: 17.51094 time: 0.43 secs
## hidden: 3, 1 thresh: 0.01 rep: 1/1 steps: 15038 error: 4.02102 time: 0.74 secs
## hidden: 2, 2 thresh: 0.01 rep: 1/1 steps: stepmax min thresh: 0.0209678873158622## Warning: Algorithm did not converge in 1 of 1 repetition(s) within the stepmax.## Warning: predictions failed for Fold5.Rep1: layer1=2, layer2=2, layer3=0 Error in cbind(1, pred) %*% weights[[num_hidden_layers + 1]] :
## requires numeric/complex matrix/vector arguments## hidden: 3, 2 thresh: 0.01 rep: 1/1 steps: 21819 error: 10.52351 time: 1.09 secs## Warning in nominalTrainWorkflow(x = x, y = y, wts = weights, info = trainInfo, : There were
## missing values in resampled performance measures.## hidden: 3 thresh: 0.01 rep: 1/1 steps: 5549 error: 22.05278 time: 0.25 secs## Neural Network
##
## 200 samples
## 19 predictor
##
## No pre-processing
## Resampling: Cross-Validated (5 fold, repeated 1 times)
## Summary of sample sizes: 160, 160, 161, 159, 160
## Resampling results across tuning parameters:
##
## layer1 layer2 RMSE Rsquared MAE
## 2 0 0.7158934 0.4777751 0.5470894
## 2 1 0.7982210 0.4647982 0.5453739
## 2 2 0.8337062 0.5028931 0.5577040
## 3 0 0.6861893 0.5651652 0.4828159
## 3 1 0.9920416 0.4133856 0.6240881
## 3 2 0.9044428 0.4639449 0.6066114
##
## Tuning parameter 'layer3' was held constant at a value of 0
## RMSE was used to select the optimal model using the smallest value.
## The final values used for the model were layer1 = 3, layer2 = 0 and layer3 = 0.Se predice su salario usando regresión lineal múltiple y neuralnet. ¿Cuál es mejor? ¿Por qué?
11.7 Redes neuronales convolucionales
Conjunto de datos de 50.000 imágenes de entrenamiento en color de 32x32, etiquetadas en más de 100 categorías y 10.000 imágenes de prueba.
Son 20 clases de mamíferos (ejemplo: acuáticos), con 5 tipos (castor, delfín, nutria, foca, ballena), 100 etiquetas en total.
Nota: el siguiente enlace tiene las instrucciones para instalar tensorflow y keras: https://tensorflow.rstudio.com/install/
# install.packages("keras")
# install_keras()
# library(tensorflow)
# install_tensorflow()
# https://tensorflow.rstudio.com/install/
library(keras3)
cifar100 <- dataset_cifar100() # Descarga/carga CIFAR-100 ya particionado en train/test
names(cifar100) # Estructura: lista con $train y $test## [1] "train" "test"##
## 0 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23
## 500 500 500 500 500 500 500 500 500 500 500 500 500 500 500 500 500 500 500 500 500 500 500 500
## 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47
## 500 500 500 500 500 500 500 500 500 500 500 500 500 500 500 500 500 500 500 500 500 500 500 500
## 48 49 50 51 52 53 54 55 56 57 58 59 60 61 62 63 64 65 66 67 68 69 70 71
## 500 500 500 500 500 500 500 500 500 500 500 500 500 500 500 500 500 500 500 500 500 500 500 500
## 72 73 74 75 76 77 78 79 80 81 82 83 84 85 86 87 88 89 90 91 92 93 94 95
## 500 500 500 500 500 500 500 500 500 500 500 500 500 500 500 500 500 500 500 500 500 500 500 500
## 96 97 98 99
## 500 500 500 500x_train <- cifar100$train$x # Tensores de imágenes: [n, 32, 32, 3]
g_train <- cifar100$train$y # Etiquetas enteras (0–99)
x_test <- cifar100$test$x
g_test <- cifar100$test$y
dim(x_train) # 50000 x 32 x 32 x 3## [1] 50000 32 32 3## [1] 13 255one_hot_encode <- function(class_vector, num_classes = NULL) {
class_vector <- as.integer(class_vector) # Asegura enteros 0..K-1
if (is.null(num_classes)) {
num_classes <- max(class_vector) + 1 # Si no dan K, se infiere K = max + 1
}
binary_matrix <- matrix(0, nrow = length(class_vector), ncol = num_classes)
for (i in seq_along(class_vector)) {
binary_matrix[i, class_vector[i] + 1] <- 1 # +1: R indexa desde 1
}
return(binary_matrix)
}
x_train <- x_train / 255 # Normaliza intensidades a [0,1] (mejora estabilidad)
x_test <- x_test / 255
y_train <- one_hot_encode(g_train, num_classes = 100) # One-hot para 100 clases
dim(y_train)## [1] 50000 100Atención:
- num_classes = 100: fija la dimensión de salida de one-hot (columnas = clases).
- Normalizar a
[0,1]ayuda a que el optimizador converja mejor.
El conjunto de 50.000 imágenes de formación tiene cuatro dimensiones:
- Cada imagen de tres colores se representa como un conjunto de tres canales, cada uno de los cuales consta de \(32\times 32\) píxeles de ocho bits.
Estandarizamos, pero manteniendo la estructura de la matriz. Codificamos la respuesta one-hot para producir una matriz binaria de 100 columnas.
Antes de comenzar, miramos algunas de las imágenes de entrenamiento usando el paquete jpeg
library(jpeg) # (opcional) utilidades de imagen
par(mar = c(0, 0, 0, 0), mfrow = c(5, 5)) # mar: márgenes; mfrow: 25 paneles (5x5)
index <- sample(seq(50000), 25) # 25 índices aleatorios del set de train
for (i in index) plot(as.raster(x_train[i,,, ])) # Renderiza 32x32x3 como imagen
La función as.raster() convierte el mapa de características (x_train) para que pueda trazarse como una imagen en color.
Aquí especificamos una CNN de tamaño moderado con fines de demostración.
# API Funcional (recomendada en keras3)
inputs <- layer_input(shape = c(32, 32, 3))
# shape: forma de una sola imagen (alto, ancho, canales). No incluye tamaño de batch.
outputs <- inputs %>%
layer_conv_2d(32, c(3,3), padding = "same", activation = "relu") %>%
# 32 filtros; kernel 3x3; padding "same" conserva tamaño espacial; ReLU como activación.
layer_max_pooling_2d(c(2,2)) %>%
# Pooling 2x2 reduce a la mitad alto/ancho.
layer_conv_2d(64, c(3,3), padding = "same", activation = "relu") %>%
layer_max_pooling_2d(c(2,2)) %>%
layer_conv_2d(128, c(3,3), padding = "same", activation = "relu") %>%
layer_max_pooling_2d(c(2,2)) %>%
layer_conv_2d(256, c(3,3), padding = "same", activation = "relu") %>%
layer_max_pooling_2d(c(2,2)) %>%
layer_flatten() %>% # Aplana [H, W, C] -> vector 1D
layer_dense(512, activation = "relu") %>% # Capa densa con 512 neuronas (no linealidad ReLU)
layer_dropout(0.5) %>% # 50% de “apagado” (regularización)
layer_dense(100, activation = "softmax") # Salida: 100 clases con softmax (probabilidades)
model <- keras_model(inputs, outputs)
model %>% compile(
optimizer = optimizer_adam(learning_rate = 1e-3),
# optimizer_adam: regla de actualización; learning_rate = 0.001 (paso inicial)
loss = "categorical_crossentropy",
# Pérdida para clasificación multiclase con labels one-hot
metrics = "accuracy"
# Reporta accuracy en entrenamiento/validación
)Parámetros de capas:
- layer_conv_2d(filters, kernel_size, padding, activation): #filtros, tamaño kernel, borde y no linealidad.
layer_max_pooling_2d(pool_size): submuestreo espacial (reduce resolución).layer_dense(units, activation): neuronas y activación (ReLU/softmax).layer_dropout(rate): proporción de neuronas “apagadas” para evitar overfitting.optimizer_adam(learning_rate = 1e-3): tamaño de paso del optimizador (ajustable).
Finalmente, especificamos el algoritmo de ajuste y ajustamos el modelo.
library(caret) # (opcional) utilidades de ML clásicas; no es requerido por Keras
library(ISLR2) # (opcional) datasets de ejemplo; no se usa aquí
# Puedes recompilar con otro optimizador para comparar (opcional):
model %>% compile(loss = "categorical_crossentropy",
optimizer = optimizer_rmsprop(), # RMSprop: paso adaptativo por dimensión
metrics = c("accuracy"))
# history <- model %>% fit(x_train, y_train, epochs = 30,
history <- model %>% fit(
x_train, y_train,
epochs = 1, # Nº de pasadas completas por el set de entrenamiento
batch_size = 128, # Tamaño del mini-lote (trade-off ruido/uso de memoria)
validation_split = 0.20 # Toma 20% del train para validación interna
)epochs: más épocas \(\approx\) mayor tiempo/posible mejor ajuste (vigilar overfitting).batch_size: 128 suele equilibrar velocidad/estabilidad; ajusta según GPU/CPU.validation_split = 0.20: separa aleatoriamente 20% dex_train/y_trainpara validar.
Nota: si quieres validar en test explícito, usa
validation_data = list(x_test, y_test)en lugar devalidation_split.
sol <- model %>% predict(x_test) # Probabilidades por clase [n_test x 100]
pred_clases <- apply(sol, 1, which.max) - 1 # Índice de argmax por fila; -1 para 0..99
# Accuracy simple (usar length(g_test), no nrow())
acc_test <- mean(pred_clases == as.integer(g_test))
acc_test## [1] 0.0966predict(x_test): devuelve probabilidades por clase.apply(..., 1, which.max): “argmax” por fila (imagen).- 1: ajusta de índice 1-based (R) a etiquetas 0-based (CIFAR-100).Corrección: usa
length(g_test)omean(...)para accuracy (nonrow(g_test)porqueg_testes vector).